來看一下圖片。這是利用不同劑量的藥劑處理非癌化細胞後,再以螢光染劑進行染色。研究者的目的是要計算這有多少細胞。
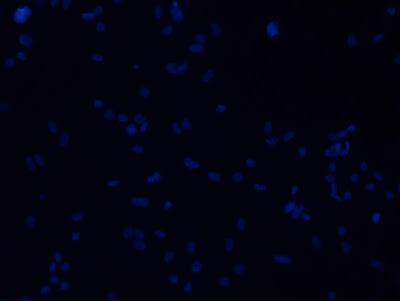
由於有些細胞是重疊的,一般想法就是直接作分水嶺切割,然後再作analyze particles。不過這樣的作法會有大問題。
這是我們期待看到的,兩個細胞被分水嶺算法剛好切開。

但實際上會發生的是,單獨一個細胞往往會被切成好幾塊

或是兩個細胞被切成三塊

如何解決這樣的問題呢?
解決流程是這樣的
(1)先把單獨未重疊的單顆細胞抓出,並計數。
(2)只針對重疊的細胞作分水嶺切割,然後作計數。
現在問題來了,怎麼知道誰是單顆細胞,這裡會運用一個形態學的運算方式來幫助判斷。
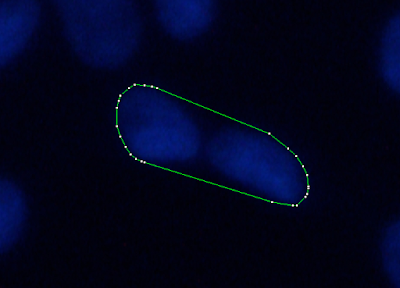
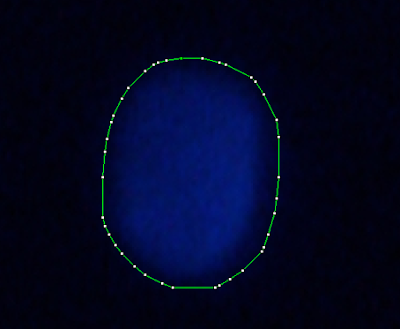
以下兩張圖是先將圖片做threshold處理後,轉二值化影像,再用wand tool選取後,然後用Edit/Selection/Convex hull做出的新選取區。
convex hull是個重點!它就像是用條橡皮筋套住你的選取區,產生的新選取區就是convex hull。你看第一張圖片,是兩個細胞重疊,用convex圈起之後,會留下很多沒圈到的地方。第二張圖片是單獨的細胞,圈起來之後留下的空白很少。
所以啊,我們可以用convex hull和細胞的面積做運算,看看細胞在hull裡頭佔面積的多寡來推論那是不是單獨的細胞。
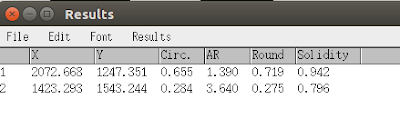
要計算這個東西,其實不用自己手動一個一個圈細胞。在measure中的測量值solidity就是這個啦。你可以勾選Analyze/Set Measurements/Shape descriptors,就可以呈現出這個數值了。
現在問題來了,怎麼知道誰是單顆細胞,這裡會運用一個形態學的運算方式來幫助判斷。
以下兩張圖是先將圖片做threshold處理後,轉二值化影像,再用wand tool選取後,然後用Edit/Selection/Convex hull做出的新選取區。
convex hull是個重點!它就像是用條橡皮筋套住你的選取區,產生的新選取區就是convex hull。你看第一張圖片,是兩個細胞重疊,用convex圈起之後,會留下很多沒圈到的地方。第二張圖片是單獨的細胞,圈起來之後留下的空白很少。
所以啊,我們可以用convex hull和細胞的面積做運算,看看細胞在hull裡頭佔面積的多寡來推論那是不是單獨的細胞。


要計算這個東西,其實不用自己手動一個一個圈細胞。在measure中的測量值solidity就是這個啦。你可以勾選Analyze/Set Measurements/Shape descriptors,就可以呈現出這個數值了。

接下來的分析和製作Macro,我就交給影片說明了
影片中所使用的Macro 就是以下的文字啦
=============================================
Dialog.create("Preference");
Dialog.addNumber("solidity:", 0.88);
Dialog.addNumber("cell area min :", 2000);
Dialog.addCheckbox("Single cell fill color", true);
Dialog.addCheckbox("Watershed cell fill color", true);
Dialog.show();
solidityValue = Dialog.getNumber();
cellAreaMin = Dialog.getNumber();
singleCheck = Dialog.getCheckbox();
watershedCheck = Dialog.getCheckbox();
//關閉所有圖檔
run("Close All");
//製作結果表
Table.create("cellCounts");
//打開資料夾,取得檔案list
dir = getDirectory("Choose a Directory ");
list = getFileList(dir);
filecount = 1;
listFiles(dir);
function listFiles(dir) {
for (i=0; i<list.length; i++) {
if (endsWith(list[i], "/")){}
// listFiles(""+dir+list[i]);
else{
//print((filecount++) + ": " + dir + list[i]);
open(dir + list[i] );
bname=File.nameWithoutExtension;
cellCounts();
}
}
}
function cellCounts(){
//細胞有幾個
cellNum_single=0; //一個單獨的細胞
cellNum_overlap=0; //重疊的細胞團
cellNum_watershed=0; //分水嶺切割後,單獨的細胞
rename("original");
//製作空白的檔案
run("Duplicate...", "title=cell_single");
run("Select All");
setBackgroundColor(255, 255, 255);
run("Clear", "slice");
run("Duplicate...", "title=cell_overlap");
run("Duplicate...", "title=cell_watershed");
//分析前處理
selectWindow("original");
run("Duplicate...", "title=a");
run("8-bit");
setAutoThreshold("Default dark");
run("Convert to Mask");
//把所有細胞都抓出,設定measure參數
run("Set Measurements...", "area perimeter shape redirect=None decimal=3");
run("Analyze Particles...", "size="+cellAreaMin+"-Infinity circularity=0.10-1.00 display exclude clear include add");
//用Solidity推斷細胞是否有重疊
numROIs = roiManager("count");
for(i=0; i<numROIs;i++) {// loop through ROIs
solidity = getResult("Solidity", i);
if(solidity>solidityValue){//Solidity大於0.88者,應該是單顆細胞
cellNum_single =cellNum_single +1;
//塗色
if (singleCheck){
selectWindow("cell_single");
roiManager("Select", i);
setForegroundColor(255*random, 255*random,255*random); //隨機填入顏色
run("Fill");
}
}
else {//如果是小於0.88,可能是多個細胞重疊,進入迴圈作分水嶺切割
cellNum_overlap =cellNum_overlap +1;
selectWindow("cell_overlap");
roiManager("Select", i);
setForegroundColor(0,0,0); //填入黑色
run("Fill");
}
}
//到cell_overlap的圖片中,執行分水嶺算法
selectWindow("cell_overlap");
run("Make Binary");
run("Watershed");
run("Select All");
run("Analyze Particles...", "size="+cellAreaMin+"-Infinity circularity=0.10-1.00 display exclude clear include add");
numROIs = roiManager("count");
for(i=0; i<numROIs;i++) {// loop through ROIs
cellNum_watershed =cellNum_watershed +1;
if(watershedCheck){
selectWindow("cell_watershed");
roiManager("Select", i);
setForegroundColor(255*random, 255*random,255*random); //隨機填入顏色
run("Fill");
}
}
//輸出細胞的計數結果
rowsize = Table.size("cellCounts");
Table.set("filename", rowsize, bname,"cellCounts");
Table.set("cell_single",rowsize, cellNum_single,"cellCounts");
Table.set("cell_overlap",rowsize, cellNum_overlap,"cellCounts");
Table.set("cell_watershed",rowsize, cellNum_watershed,"cellCounts");
Table.update("cellCounts");
//關閉圖檔
selectWindow("cell_single");
saveAs("Jpeg", dir+"1/" + bname+"_1_single"+".Jpg");
selectWindow("cell_overlap");
saveAs("Jpeg", dir+"1/" + bname+"_2_overlap"+".Jpg");
selectWindow("cell_watershed");
saveAs("Jpeg", dir+"1/" + bname+"_3_watershed"+".Jpg");
close("cell_single");
close("cell_overlap");
close("cell_watershed");
close("a");
close("original");
close("Results");
close("ROI Manager");
}

