前幾天看到小p拿出他的兵器-做水波干涉實驗的兵器,錄製了一段影片。突然我想到我在這篇《imagej將時間資料視覺化》寫到imagej可以將時間資料濃縮,可以從中看出特別的時間資訊。
一時手癢就來把小p拍攝的影片用imagej疊圖分析看看。
1.先用youtube-dl把影片用mp4格式下載回來。
2.用fiji(特製的imagej)的file/import/Movie(FFMPEG)將mp4檔讀成stacks
3.用Image/Stacks/Z Project製作疊圖
以下分別是用Min intensity和standard deviation製作的疊圖
Min intensity是把影像中每個隨著時間變化的像素,用最小的數值(相當於最暗的)去疊出來。
而standard deviation的圖我是第一次用這疊,發現還蠻有趣的。疊出來的影像中,黑色的部份代表標準差很小,意思就是數值變動不大,所以在影片中沒什麼變化的點就會呈現黑色。而動得越厲害的部份,就會越彩色。所以左方的水波產生器顏色就很彩色。
把影片做成疊圖來分析,在看水波干涉時產生的腹線(antinodal line),如中央腹線、第一腹線、第二腹線等可是特別明顯呢。
2018年10月30日
2018年10月28日
使用imagej的Macro切割細胞與細胞追蹤
來自網友的這個提問,其實目的是要做細胞追蹤,要處理的是細胞分裂的影片。希望能將A細胞在每個影格都能定位出來,網友也提供了判斷邏輯。將第一影格的A細胞和第二影格的兩個細胞都做交集處理,看看和第二影格的哪個細胞交集的比較多,就可以找到第二影格的A細胞在哪。
由於之前沒有寫過類似的Macro,所以也看了幾次說明文件,還參考了其他人寫的Macro,總算是寫出來了。要處理這個問題之前,其實得先把細胞切割好,還好網友已經先切好了給我處理。
影片中用的Macro我貼在本文最後,使用前提是要用wand tool先點選一個細胞,加入ROI Manager之後,再去執行Macro。
雖然網友已經提供給我切割好的細胞輪廓,不過我也還是手癢想處理看看這個影片,想說有沒有辦法可以把細胞切割好,不過我試了好幾個方法都切不好。最後只好用半自動的方法來做。
方法是先手動在細胞分界處劃線,存成ROI,再用這個ROI去切割開細胞。具體的作法就看影片囉
*********************************
setBatchMode(true);
setAutoThreshold("Default");
width = getWidth;
height = getHeight;
n = getSliceNumber();
for (i=n; i<=nSlices-1; i++) {
//for (i=n; i<=n; i++) {
print("==========");
print("i=",i);
roiMan_n = roiManager("count");
print("roiMan_n=",roiMan_n);
run("Select None");
setSlice(i+1);
run("Analyze Particles...", "display add slice");
roiMan_n_new = roiManager("count");
print("roiMan_n_new=",roiMan_n_new);
//計算下一張Slice增加了多少個ROI
roi_add = roiMan_n_new - roiMan_n ;
print("roi_add=",roi_add);
roiManager("Show None");
AND_Big_area= 0; //預設取交集後的面積為零
AND_Big_area_n = 0 ;//預設取交集後的面積,最大的是第零個
for (j=1; j<=roi_add; j++) {
roiManager("Select", newArray(roiMan_n-1,roiMan_n-1+j));
roiManager("AND");
getStatistics(area);
print("j=",j,",area=",area);
//沒交集的面積會回報成整張圖片的面積,所以將回報面積設為0
if(area == width * height){
area =0;
}
if(area>AND_Big_area){
AND_Big_area = area;
AND_Big_area_n = j;
}
}
print("Big=",AND_Big_area_n);
print("AND_Big_area=",AND_Big_area);
//select biggest area and add the new ROI in the next slice
roiManager("Select", roiMan_n -1 + AND_Big_area_n);
roiManager("Add");
//delete all new ROI in the next slice
for (j=1; j<=roi_add; j++) {
roiManager("Select", roiMan_n-1+1);
roiManager("Delete");
}
}
resetThreshold();
setBatchMode(false);
使用imagej將影像中的維管束群組化
最近一個網友來信問到的問題,是和影像群組化有關的。這方向的問題也是很多提問的人會問到的,不如在影片中詳細解說一下,有此疑惑的也可以藉此練習看看。
就以下面這圖為例,大方塊和小方塊在做Analyze Particles時,會被視作是不同的兩個Particle,但是如果我們的需求是要把這兩個圈在同一個ROI,那應該怎麼做?
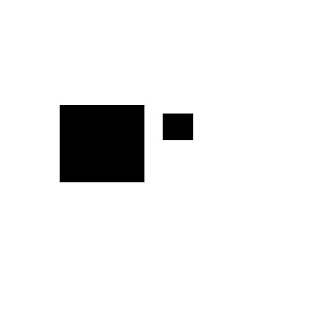
什麼時候會有這種情況呢?來看看這個例子,這是竹子維管束的圖片,右圖是進行threshold之後,維管束分離的樣子,而在分析的時候,我們需要將那些分離的小碎片視作是一個完整的Particle。

以下四部影片就是在介紹這些步驟
第一部影片:imagej 將分散的小區塊用二值化影像處理流程做群組1 二值化處理的功能說明
第二部影片:imagej 將分散的小區塊用二值化影像處理流程做群組2 一步一步說明
利用上述的二值化程序一步一步說明,如何將影像中分散的小顆粒在分析時處理成一個完整的部份。
第三部影片:imagej 將分散的小區塊用二值化影像處理流程做群組3 用程式碼加快處理流程
將第二部的步驟化處理程序寫成Imagej的Macro程式碼。程式碼的檔案我放在雲端硬碟,文字我也貼在本文的最後。
就以下面這圖為例,大方塊和小方塊在做Analyze Particles時,會被視作是不同的兩個Particle,但是如果我們的需求是要把這兩個圈在同一個ROI,那應該怎麼做?
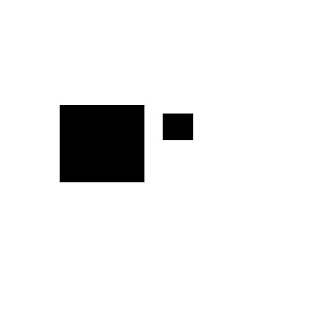
什麼時候會有這種情況呢?來看看這個例子,這是竹子維管束的圖片,右圖是進行threshold之後,維管束分離的樣子,而在分析的時候,我們需要將那些分離的小碎片視作是一個完整的Particle。

以下四部影片就是在介紹這些步驟
第一部影片:imagej 將分散的小區塊用二值化影像處理流程做群組1 二值化處理的功能說明
是進行二值化處理的教學流程,介紹erode、dilate等程序的作用
第二部影片:imagej 將分散的小區塊用二值化影像處理流程做群組2 一步一步說明
利用上述的二值化程序一步一步說明,如何將影像中分散的小顆粒在分析時處理成一個完整的部份。
將第二部的步驟化處理程序寫成Imagej的Macro程式碼。程式碼的檔案我放在雲端硬碟,文字我也貼在本文的最後。
第四部影片:imagej用Macro將影像的ROI都各自存圖
將分析後的ROI各自存成圖,然後再組成一個Montage
將分析後的ROI各自存成圖,然後再組成一個Montage
結果會如這張圖
程式碼比較簡單一些,我直接貼在這邊
***********************
path = "/home/shawn-pc/桌面/crop/";
//path="c:/users/user/Desktop/crop/";
fileTitle=getTitle;
for (i=0; i<roiManager("count"); ++i) {
roiManager("Select", i);
roiname = call("ij.plugin.frame.RoiManager.getName", i);
run("Duplicate...", "title=crop");
saveAs("Jpeg", path + roiname +".jpeg");
close();
//Next round!
selectWindow(fileTitle);
}
***********************
第三部影片提到的程式碼,則是這樣
*************************
//input = "C:/Users/user/Desktop/roi/";
input = "/home/shawn-pc/桌面/imagej竹子維管束/roi/"
particle_size = "70-140";
particle_size_connect= "150-500";
particle_size_horseshoe = "30-200";
particle_size_fragment = "10-100";
particle_cir = "0.6-1.0";
particle_cir_01 = "0.0-1.0";
analy_string = "size="+ particle_size +" circularity="+ particle_cir +" show=Masks clear include summarize add";
analy_string_connect = "size="+ particle_size_connect +" circularity= " + particle_cir_01 +" show=Masks clear include summarize add";
analy_string_horseshoe = "size="+ particle_size_horseshoe +" circularity= " + particle_cir_01 +" show=Masks clear include summarize add";
analy_string_fragment = "size="+ particle_size_fragment +" circularity= " + particle_cir_01 +" show=Masks clear include summarize add";
function single(m){
run("Duplicate...", "title="+m);
run("Analyze Particles...", analy_string);
if(Table.size != 0){
//if ((Table.get("Count",Table.size-1)) != 0) {
roiManager("Save", input + m +".zip");
selectWindow("0");
n=roiManager("count");
for(i=0;i<n;i++){
roiManager("Select", i);
setForegroundColor(255, 255, 255);
run("Fill", "slice");
}
}
n=roiManager("count");
for(i=0;i<n;i++){
roiManager("Select", 0);
roiManager("delete");
}
run("Select None");
roiManager("show none");
close("Mask of "+m);
close("Mask of 0");
close("Mask of Mask of 0");
close(m);
}
function connect(m){
run("Analyze Particles...", analy_string_connect);
run("Watershed");
single(m);
}
//=========================================
name=getTitle;
run("Duplicate...", "title=0");
setAutoThreshold("Default");
run("Convert to Mask");
single(1);
connect(2);
//抓馬蹄形,單獨的
run("Analyze Particles...", "size=70-140 circularity=0.00-1.00 show=Masks clear include summarize add");
run("Close-");
single(3);
//抓馬蹄形,連在一起的
run("Analyze Particles...", analy_string_horseshoe);
run("Close-");
connect(4);
//抓碎片
run("Analyze Particles...", analy_string_horseshoe);
run("Close-");
single(5);
//抓更碎片
run("Analyze Particles...", analy_string_fragment);
run("Close-");
run("Dilate");
run("Fill Holes");
run("Watershed");
run("Erode");
single(6);
//抓更更碎片
run("Analyze Particles...", analy_string_fragment);
run("Dilate");
run("Fill Holes");
run("Erode");
single(7);
list = getFileList(input);
for (i = 0; i < list.length; i++){
roiManager("Open", input+list[i]);
}
selectWindow(name);
roiManager("show none");
roiManager("Show All without labels");
*************************
2018年10月12日
用在生態系的AR App介紹: WWF Free Rivers
昨天看到Apple網站上對新版本的iPad的廣告,特別強調了AR擴增實境的介紹。特別點進去看,看到了介紹幾款APP,然後我就試用了這個WWF Free Rivers。
這是WWF世界自然基金會出品的,簡單地說這個APP是個用AR方式帶你看一個模型裡發生的故事,本身互動性我覺得還好,沒有什麼特別的互動功能,但是能夠你讓你用上帝的視角去東看西看。
APP一開始先讓你設定模型的位置,你需要一張桌子(或是地板),然後你就決定模型要擺放的方向。這APP有中文介面,也有中文介紹,畫面就如同下面這樣,是個以河流為主的模型。不過陸域的設定其實是綜合的,看那裏面的動物,同時有熊、獅子、老虎、鱷魚還有白鱀豚...。大概是包含了非洲草原、熱帶雨林、三角洲等等之類的。
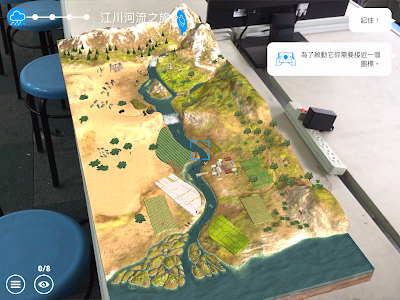
可以看看河流和周圍動物的之間的關係,或是人類怎麼利用河流,然後故事會進展到看到起雲降雨(這個場景用AR來看還蠻有意思的),接著就是蓋起水壩,看看水壩上游和下游有什麼不同,然後就是拆掉水壩再看看有什麼變化。蓋水壩是為了能源需求,所以場景中也加入了可以用風力發電來提供能源。

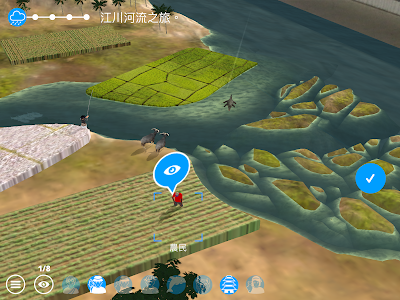

除了上述的場景外,也提供另外一個體驗,它像是你面向一個大展版,展板前方還有一些模型這樣。


我覺得這個APP在生態和環境的課程中應該可以使用看看,不過就不適合老師絮絮叨叨地介紹,應該是提供一個觀察提問的指引單,讓學生透過模型去看那些現象。
這是WWF世界自然基金會出品的,簡單地說這個APP是個用AR方式帶你看一個模型裡發生的故事,本身互動性我覺得還好,沒有什麼特別的互動功能,但是能夠你讓你用上帝的視角去東看西看。
APP一開始先讓你設定模型的位置,你需要一張桌子(或是地板),然後你就決定模型要擺放的方向。這APP有中文介面,也有中文介紹,畫面就如同下面這樣,是個以河流為主的模型。不過陸域的設定其實是綜合的,看那裏面的動物,同時有熊、獅子、老虎、鱷魚還有白鱀豚...。大概是包含了非洲草原、熱帶雨林、三角洲等等之類的。

可以看看河流和周圍動物的之間的關係,或是人類怎麼利用河流,然後故事會進展到看到起雲降雨(這個場景用AR來看還蠻有意思的),接著就是蓋起水壩,看看水壩上游和下游有什麼不同,然後就是拆掉水壩再看看有什麼變化。蓋水壩是為了能源需求,所以場景中也加入了可以用風力發電來提供能源。



除了上述的場景外,也提供另外一個體驗,它像是你面向一個大展版,展板前方還有一些模型這樣。


我覺得這個APP在生態和環境的課程中應該可以使用看看,不過就不適合老師絮絮叨叨地介紹,應該是提供一個觀察提問的指引單,讓學生透過模型去看那些現象。
2018年10月8日
用QGIS取出海岸線的方法
最近一個網友來信問了問題,他想跟著做這篇文章的東西。
QGIS作透明的立體等高線地形模型
QGIS作透明的立體等高線地形模型
但是有個問題,取等高線的時候沒辦法直接取出等高線為0的線,其實也就是海岸線。
取不出那條海岸線的原因其實是因為圖檔中數值為0的地方為海,並不是海岸線。意思就是如果我們光看圖檔裡的數值,其實海洋的區域就是滿滿的0,而海岸線其實是一堆零和非零的數字交界的邊緣。
所以要取出海岸線就要換個想法來做,首先把所有有高度的地方(非零)都變成1,也就是做成二值化影像,然後將影像向量化,就可以取出海岸線的那條線了。
做法我也錄成影片啦
訂閱:
意見 (Atom)


