最近有人在問我,想要用ImageJ來測物體的最大粒徑和最小粒徑。
我先以最大粒徑來說明,因為這直接用Imagej的功能就可以了,不用加外掛。
假設你要測的是這個血液抹片裡紅血球的平均最大粒徑,先設定
Analyze/Set Measurements...
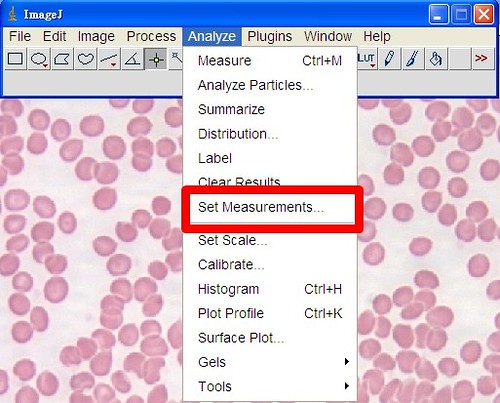
把Feret's Diameter 打開
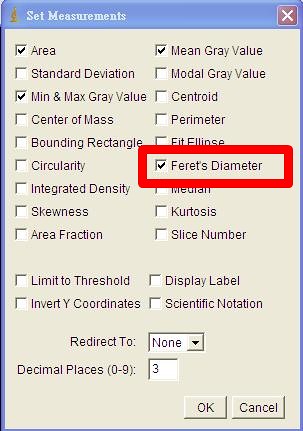
紅血球圖片作二位元的處理(記得事前要先作比例尺校正,我這個例子省略了這部份)
Process/Binary/Make Binary
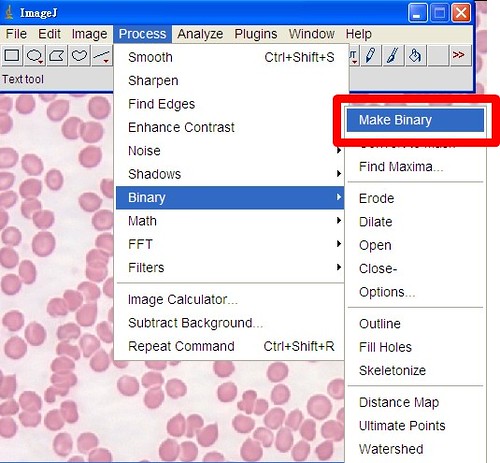
變成黑白之後,再分析粒子
Analyze/ Analyze Particles...
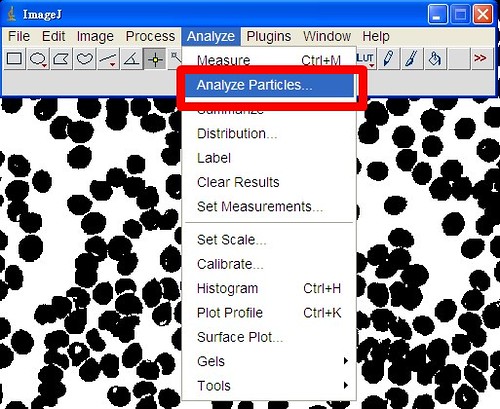
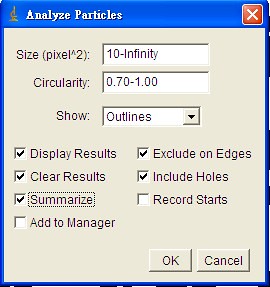
Size 我設定由10-無限大,所以面積在10以下的小顆粒忽略不計。這裡要設定多少,得看你圖的性質而定。
Circularity,設定0.70-1.00,所以不夠圓的(0.6以下)不估計。這是因為照片裡有很多紅血球都重疊在一起,如果連那些一起算,那又會高估最大粒徑,所以我只算那些獨立存在的紅血球。
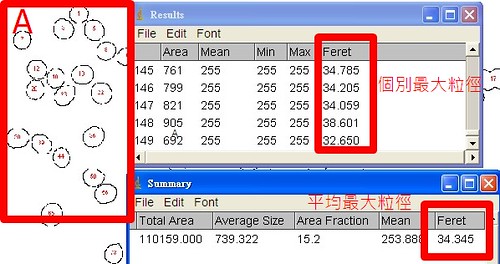
分析之後的三個視窗,左邊那個A是被分析的樣本,有納入的都有被框起來,並且被標號。
Results裡是149顆紅血球的各個最大粒徑,summary裡是平均的最大粒徑。(注意:這個沒有作校正,所以這裡的長度只是像素值)
如果你是要手動作計算,你要花多久時間?不過用Imagej來作,不用30秒就完成了。
更新:不知道哪一版開始,Imagej的Feret已經有測量最小徑的功能了,都是用以上同樣的方式去設定,最後在Results就會有MinFeret的欄位,那就是最小徑。所以不需要再裝以下的外掛了,其實底下的外掛都已經掛掉了,所以你要裝也是沒辦法裝的。
如果你除了最大粒徑以外,還要計算最小值,那麼就要加Rotating Callipers這個外掛了。
一個是可以安裝這裡的macros
http://doube.org/macros.html
或是這裡的plugins
http://doube.org/plugins.html
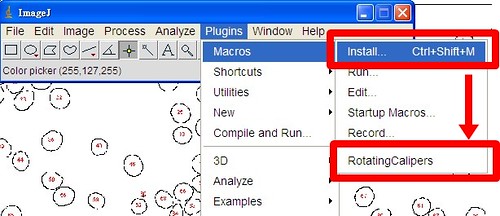
我用macros來說明,到上面的網頁把macros下載到電腦,它是個文字檔。
安裝方式:plugins/install
這樣在底下就會出現一個Rotating Callipers
使用方式,一種是把你要分析的粒子或細胞,一個一個用點選工具累積選取。然後去選擇Rotating Callipers。
另外一種是在前面的分析過程中,在下面這個步驟把左下角的Add manager打勾

分析完會出現ROI manager,到ROI manager把裡頭出現的那些的粒子都選取,點下Rotating Callipers,這樣就會出現最大粒徑和最小粒徑的值了。

我先以最大粒徑來說明,因為這直接用Imagej的功能就可以了,不用加外掛。
假設你要測的是這個血液抹片裡紅血球的平均最大粒徑,先設定
Analyze/Set Measurements...

把Feret's Diameter 打開

紅血球圖片作二位元的處理(記得事前要先作比例尺校正,我這個例子省略了這部份)
Process/Binary/Make Binary

變成黑白之後,再分析粒子
Analyze/ Analyze Particles...

Size 我設定由10-無限大,所以面積在10以下的小顆粒忽略不計。這裡要設定多少,得看你圖的性質而定。
Circularity,設定0.70-1.00,所以不夠圓的(0.6以下)不估計。這是因為照片裡有很多紅血球都重疊在一起,如果連那些一起算,那又會高估最大粒徑,所以我只算那些獨立存在的紅血球。

分析之後的三個視窗,左邊那個A是被分析的樣本,有納入的都有被框起來,並且被標號。
Results裡是149顆紅血球的各個最大粒徑,summary裡是平均的最大粒徑。(注意:這個沒有作校正,所以這裡的長度只是像素值)

如果你是要手動作計算,你要花多久時間?不過用Imagej來作,不用30秒就完成了。
更新:不知道哪一版開始,Imagej的Feret已經有測量最小徑的功能了,都是用以上同樣的方式去設定,最後在Results就會有MinFeret的欄位,那就是最小徑。所以不需要再裝以下的外掛了,其實底下的外掛都已經掛掉了,所以你要裝也是沒辦法裝的。
一個是可以安裝這裡的macros
http://doube.org/macros.html
或是這裡的plugins
http://doube.org/plugins.html
我用macros來說明,到上面的網頁把macros下載到電腦,它是個文字檔。
安裝方式:plugins/install
這樣在底下就會出現一個Rotating Callipers

使用方式,一種是把你要分析的粒子或細胞,一個一個用點選工具累積選取。然後去選擇Rotating Callipers。
另外一種是在前面的分析過程中,在下面這個步驟把左下角的Add manager打勾

分析完會出現ROI manager,到ROI manager把裡頭出現的那些的粒子都選取,點下Rotating Callipers,這樣就會出現最大粒徑和最小粒徑的值了。
