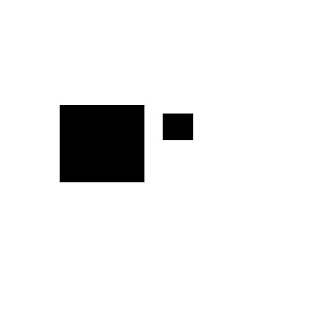最近一個網友來信問到的問題,是和影像群組化有關的。這方向的問題也是很多提問的人會問到的,不如在影片中詳細解說一下,有此疑惑的也可以藉此練習看看。
就以下面這圖為例,大方塊和小方塊在做Analyze Particles時,會被視作是不同的兩個Particle,但是如果我們的需求是要把這兩個圈在同一個ROI,那應該怎麼做?
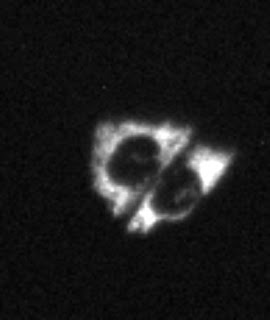
什麼時候會有這種情況呢?來看看這個例子,這是竹子維管束的圖片,右圖是進行threshold之後,維管束分離的樣子,而在分析的時候,我們需要將那些分離的小碎片視作是一個完整的Particle。
以下四部影片就是在介紹這些步驟
第一部影片:imagej 將分散的小區塊用二值化影像處理流程做群組1 二值化處理的功能說明
是進行二值化處理的教學流程,介紹erode、dilate等程序的作用
VIDEO
第二部影片:imagej 將分散的小區塊用二值化影像處理流程做群組2 一步一步說明
利用上述的二值化程序一步一步說明,如何將影像中分散的小顆粒在分析時處理成一個完整的部份。
VIDEO
第三部影片:imagej 將分散的小區塊用二值化影像處理流程做群組3 用程式碼加快處理流程
將第二部的步驟化處理程序寫成Imagej的Macro程式碼。
程式碼的檔案我放在雲端硬碟 ,文字我也貼在本文的最後。
VIDEO
第四部影片:imagej用Macro將影像的ROI都各自存圖
VIDEO
結果會如這張圖
程式碼比較簡單一些,我直接貼在這邊
***********************
path = "/home/shawn-pc/桌面/crop/";
//path="c:/users/user/Desktop/crop/";
fileTitle=getTitle;
for (i=0; i<roiManager("count"); ++i) {
roiManager("Select", i);
roiname = call("ij.plugin.frame.RoiManager.getName", i);
run("Duplicate...", "title=crop");
saveAs("Jpeg", path + roiname +".jpeg");
close();
//Next round!
selectWindow(fileTitle);
}
***********************
第三部影片提到的程式碼,則是這樣
*************************
//input = "C:/Users/user/Desktop/roi/";
input = "/home/shawn-pc/桌面/imagej竹子維管束/roi/"
particle_size = "70-140";
particle_size_connect= "150-500";
particle_size_horseshoe = "30-200";
particle_size_fragment = "10-100";
particle_cir = "0.6-1.0";
particle_cir_01 = "0.0-1.0";
analy_string = "size="+ particle_size +" circularity="+ particle_cir +" show=Masks clear include summarize add";
analy_string_connect = "size="+ particle_size_connect +" circularity= " + particle_cir_01 +" show=Masks clear include summarize add";
analy_string_horseshoe = "size="+ particle_size_horseshoe +" circularity= " + particle_cir_01 +" show=Masks clear include summarize add";
analy_string_fragment = "size="+ particle_size_fragment +" circularity= " + particle_cir_01 +" show=Masks clear include summarize add";
function single(m){
run("Duplicate...", "title="+m);
run("Analyze Particles...", analy_string);
if(Table.size != 0){
//if ((Table.get("Count",Table.size-1)) != 0) {
roiManager("Save", input + m +".zip");
selectWindow("0");
n=roiManager("count");
for(i=0;i<n;i++){
roiManager("Select", i);
setForegroundColor(255, 255, 255);
run("Fill", "slice");
}
}
n=roiManager("count");
for(i=0;i<n;i++){
roiManager("Select", 0);
roiManager("delete");
}
run("Select None");
roiManager("show none");
close("Mask of "+m);
close("Mask of 0");
close("Mask of Mask of 0");
close(m);
}
function connect(m){
run("Analyze Particles...", analy_string_connect);
run("Watershed");
single(m);
}
//=========================================
name=getTitle;
run("Duplicate...", "title=0");
setAutoThreshold("Default");
run("Convert to Mask");
single(1);
connect(2);
//抓馬蹄形,單獨的
run("Analyze Particles...", "size=70-140 circularity=0.00-1.00 show=Masks clear include summarize add");
run("Close-");
single(3);
//抓馬蹄形,連在一起的
run("Analyze Particles...", analy_string_horseshoe);
run("Close-");
connect(4);
//抓碎片
run("Analyze Particles...", analy_string_horseshoe);
run("Close-");
single(5);
//抓更碎片
run("Analyze Particles...", analy_string_fragment);
run("Close-");
run("Dilate");
run("Fill Holes");
run("Watershed");
run("Erode");
single(6);
//抓更更碎片
run("Analyze Particles...", analy_string_fragment);
run("Dilate");
run("Fill Holes");
run("Erode");
single(7);
list = getFileList(input);
for (i = 0; i < list.length; i++){
roiManager("Open", input+list[i]);
}
selectWindow(name);
roiManager("show none");
roiManager("Show All without labels");
*************************